RNA-Polimerasi
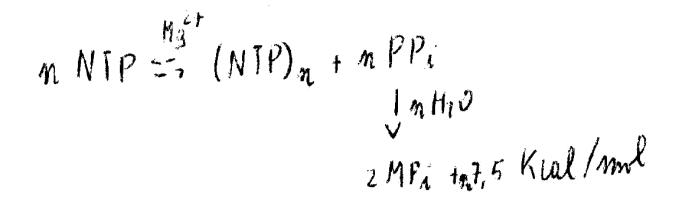
Si prende come modello la struttura dei procarioti.
C’è un’unica proteina con 3 domini strutturali. Negli eucarioti ogni dominio è formato da un protomero.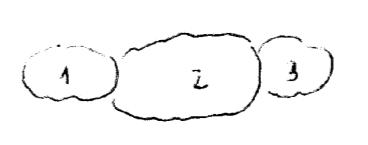
Il secondo dominio è α-GIRO-α, negli eucarioti si arriva ad un aggregato quaternario.
Il primo e il terzo dominio sono β-GIRO-β, negli eucarioti sono subunità indipendenti a struttura quaternaria.
Il secondo dominio è un sito catalitico tubolare, il cui solco è accessibile dall’esterno quando la proteina passa da conformazione tesa e chiusa a rilassata. Il primo e il terzo dominio sono siti catalitici a cavità, con organizzazione più sempplice.
I sistemi polimerasici si attivano quando si legano a molecole segnale (secondi messaggeri ormonali; messaggeri legati a cambiamenti del microambiente).
Il sito catalitico tubolare, che si estende per tutto il secondo dominio, quando la proteina passa da tesa a rilassata, può ospitare un filamento singolo. Per il DNA è necessario che ci sia un filamento di DNA singolo in modo da poter essere accettato. Il DNA è molto protetto dalle proteine: è quindi richiesto l’intervento di proteine che ricoprono il DNA, unendosi reversibilmente attraverso interazioni deboli, con la formazione di complessi. Si verifica un aumento della costante di dissociazione verso il DNA, che inizia a privarsi delle proteine di copertura. Il DNA comincia a interagire con le proteine enzimatiche idrolasi che promuovono tagli singoli o simmetrici reversibili. Il DNA cambia orientamento e si disavvolge; i superavvolgimenti si risolvono e le idrolasi intervengono.
Le proteine a dita di Zn o a cerniera di leucina si inseriscono nei solchi, e con interazioni di appaiamento promuovono lo srotolamento della doppia elica. SI formano quindi filamenti singoli mantenuti da proteine ausiliarie. I filamenti vengono riconosciuti dalle polimerasi, che si posizionano in modo da accettare catene che serviranno da stampo. La catena viene riconosciuta a partire da sequenze ripetitive di T e A. L’orientamento di lettura è nel senso 3’-5’. Per avere inizio di polimerizzazione occorre che vi sia una catena preformata (poliribonucleotidi, T viene sostituita da U): primer.
I primers offrono il primo punto di aggancio per il polimero in costruzione. Solo dopo il posizionamento del primer e dello stampo la proteina può accettare i nucleotidi. Questi entrano uno per volta secondo la complementarietà che hanno con lo stampo. Si realizza un appaiamento tra basi e posizionamento.
Mg ha interazioni tra αP e βP, quindi si forma un legame covalente. OH in 3’ dà attacco nucleofilo su P cationico: il primo nucleotide è stato agganciato. Mg torna al rapporto di legame dativo col pirofosfato, che viene sequestrato da una pirofosfatasi. L’energia derivata dalla pirofosfatasi è usata per un cambiamento conformazionale, con il risultato finale di permettere l’entrata del secondo nucleotide.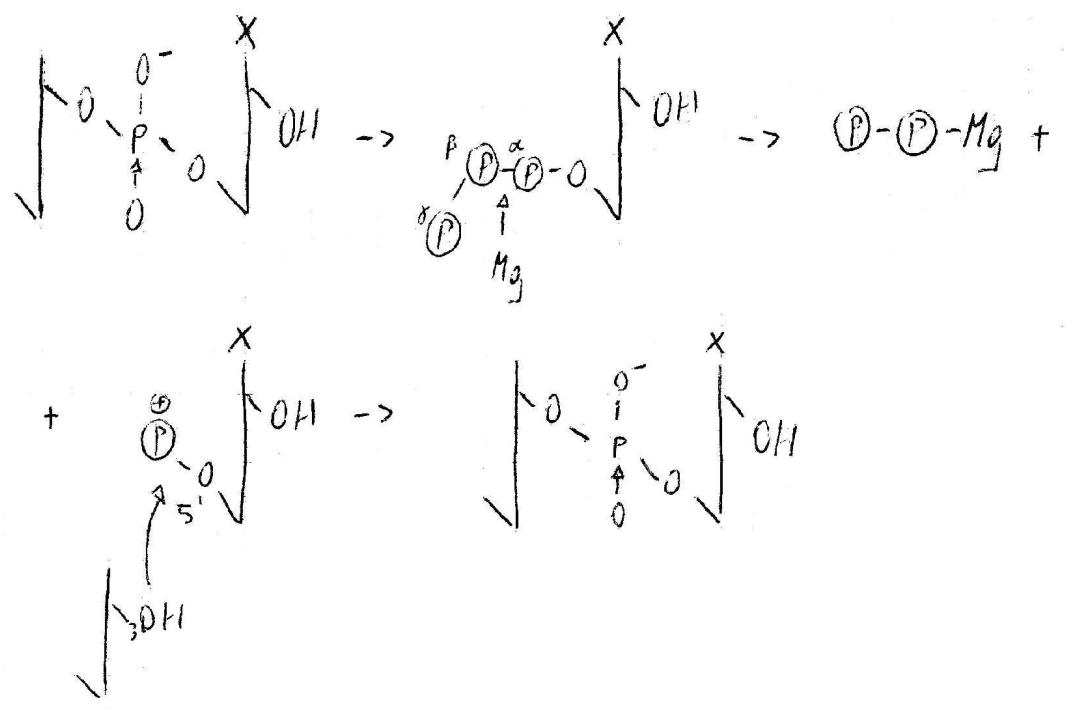
Il primo dominio ha attività idrolasica (ribonucleotide esonucleasi). Riconosce il primer nella subunità 5’ e lo digerisce fermandosi al primo desossiribonucleotide.
Il terzo dominio è una esonucleasi che riconosce le catene senza struttura attraverso il terminale 3’.
Il primo e il terzo dominio sono correttori di bozze. Normalmente l’ingresso dei nucleotidi è sempre molto preciso, ma possono succedere errori d’inserzione. Il cambiamento di conformazione dovuto al pirofosfato fa retrocedere la catena di una posizione, con il nucleotide non corretto che si trova ad essere sulla terza subunità. Viene riconosciuto il terminale 3’, si ha destabilizzazione e il nucleotide errato viene eliminato. A questo punto la polimerasi riprende correttamente.
Il DNA esiste come duplex, quindi la reazione polimerasica coinvolge le due catene (orientazione antiparallela). Vi è il problema della lettura che va da 3’ a 5’, dato che le due catene sono copiate in modo quasi simultaneo.
Una catena (filamento veloce) segue lo schema precedente. La polimerasi può passare per traslazione da una catena all’altra cambiando senso di lettura. Ad ogni passaggio però ha bisogno di un primer. Il filamento veloce viene prodotto in continuo (c’è sempre OH in 3’ disponibile).
La seconda catena è prodotta in maniera discontinua perché ha sempre bisogno del primer. Al termine si ha il filamento veloce con la parte iniziale digerita dal primer. Si ha anche il secondo filamento formato a segmenti: si hanno quindi incisioni nei filamenti. Vi sono allora altre polimerasi che con lo stesso metodo riempiono le incisioni con i nucleotidi giusti. Nel filamento veloce c’è un solo punto di discontinuità (più parti sono state digerite). Interviene allora una ligasi che salda i nucleotidi mancanti.
La duplicazione del DNA è definita anche come sintesi semiconservativa. Al termine del processo si hanno due catene duplex: quella vecchia che ha fatto da stampo e quella appena sintetizzata.
Nei DNA lineari c’è un solo punto in cui si ha la concentrazione di nucleotidi in cui può inserirsi il primer, nei DNA circolari può essercene uno o due simmetrici.
Continua a leggere:
- Successivo: Trascrizione
- Precedente: DNA-Polimerasi
Dettagli appunto:
-
Autore:
Marco Lazzara
[Visita la sua tesi: "Determinazione di arsenico mediante voltammetria di stripping anodico con elettrodo in oro"]
[Visita la sua tesi: "I laser a coloranti: fondamenti e stato dell'arte"]
- Università: Università degli Studi di Torino
- Facoltà: Scienze Matematiche, Fisiche e Naturali
- Corso: Chimica
- Esame: Biochimica
- Docente: Carlo Giunta
Altri appunti correlati:
- Biochimica
- Fondamenti di chimica
- Appunti di Biochimica
- Catecolamine e glucagone
- Chimica Analitica Strumentale (Spettroscopia)
Per approfondire questo argomento, consulta le Tesi:
- Analisi dei nitriti attraverso il loro impiego nell’industria alimentare e applicazione di un’indagine percettiva sui consumatori
- Meccanismi biochimici ed effetti sulla salute umana del licopene
- Studio dei processi di degrado su manufatti archeologici in ambiente marino sommerso e sperimentazione di procedure innovative per la conservazione in situ
- Bio-lubricant from the olive mill waste through a new biocatalyst immobilized on ZnFe2O4
- Rational Design and Synthesis of Novel Acadesine-Like Modulators of AMPK
Puoi scaricare gratuitamente questo appunto in versione integrale.