Codificazione genetica e sintesi proteica
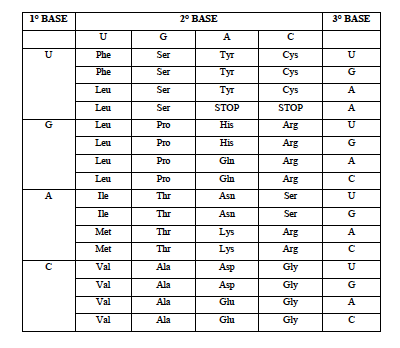
Il codice è ridondante: ci sono più possibilità per lo stesso amminoacido. Met e Trp hanno invece un solo codice.
STOP: segnale di arresto e fine della costruzione della sequenza peptidica.
I ribosomi hanno una superficie di contatto e una superficie idrodinamica (contatto col mezzo acquoso), dove sporgono le basi per il riconoscimento verso enzimi o verso strutture che portano a struttura quaternaria.
Il ribosoma ha affinità per l’RNA, mentre il DNA ne ha poca, quindi la trascrizione DNA → RNA per iniziare i processi di sintesi peptidica di sintesi peptidica deve avvenire attraverso i ribosomi.
IF1, IF2, IF3: FATTORI DI INIZIO
EFTn, EFTS, EFG: FATTORI DI ALLUNGAMENTO (TRANSFERASI)
RF1, RF2, RF3: FATTORI DI RILASCIO.
Collaborano alla sintesi peptidica, partendo dall’informazione trasmessa, con meccanismo transferasico attraverso GTP, composto ad alta energia. Per realizzare la sintesi della proteina, il ribosoma deve dissociarsi nelle 2 subunità. Si ha un aumento della costante di dissociazione dovuta a cambiamenti conformazionali dovuti alla disponibilità dei fattori d’inizio che si associano ai siti della subunità minore.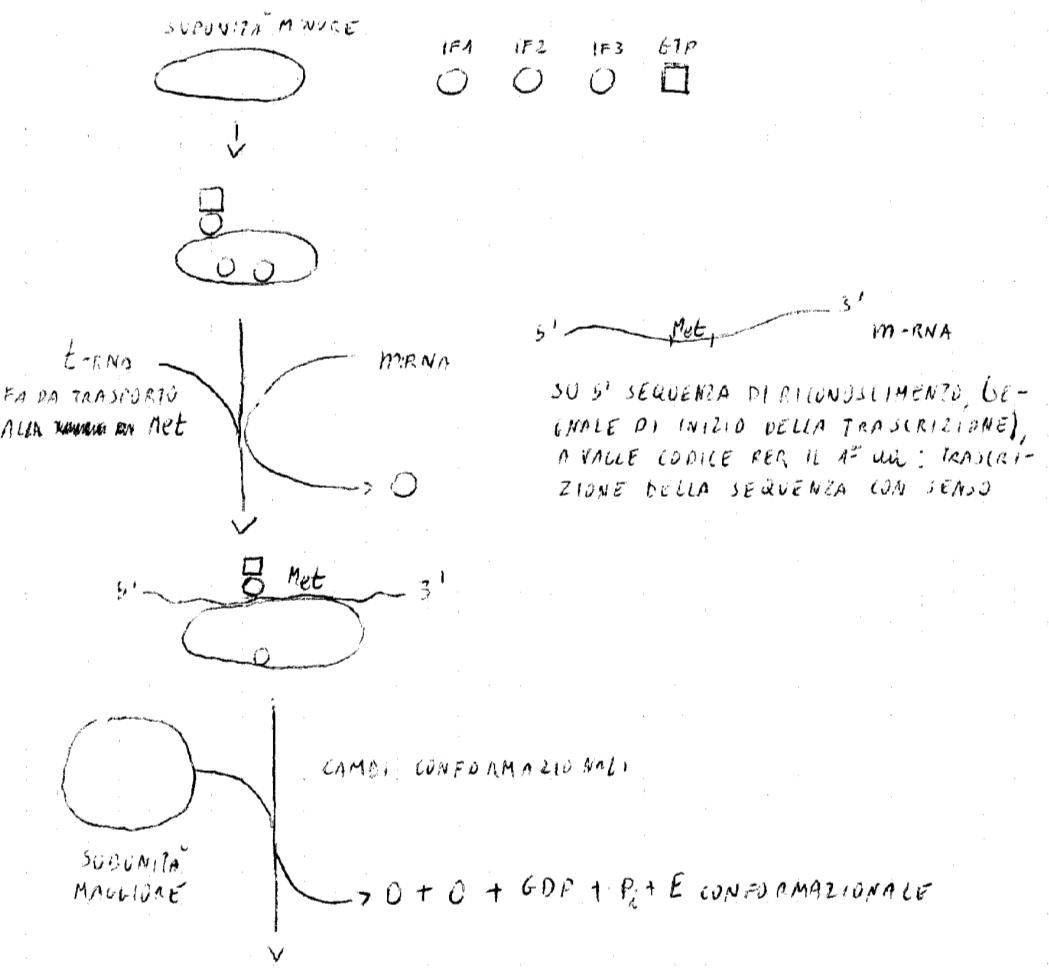
Le zone superficiali dell’unità maggiore individuano dei siti in grado di essere riconosciuti e di essere riconosciuti e di riconoscere altri partecipanti alla sintesi proteica: gli enzimi e t-RNA, che vengono riconosciuti attraverso la sequenza delle due anse e l’anticodice. L’RNA sul ribosoma contiene il codice, mentre i siti della subunità maggiore consentono il riconoscimento della proteina.
SUBUNITÁ MAGGIORE: 2 SITI (A, P) O 3 SITI (A, P, U)
• SITO A: t-RNA I aa (SITO DELL’AMMINOACIDO)
• SITO P: t-RNA II aa (SITO DEL PEPTIDE)
• SITO U (SITO DI USCITA)
Non sono entità fisse, ma definite in base alla costante di dissociazione verso il ligando specifico.
• SITO A: COSTANTE PIÚ BASSA, AFFINITÁ PIÚ ALTA
• SITO P: COSTANTE INTERMEDIA
• SITO U: COSTANTE PIÚ ALTA, RILASCIO IMMEDIATO DEL LIGANDO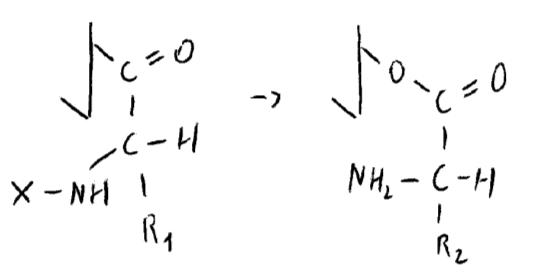
GTP → GDP + Pi
Il sito A è il sito per il 1° t-RNA di riconoscimento. Il sito P è il sito a cui si lega il 2° t-RNA col 2° amminoacido. Dev’essere riconosciuto da A, ma per il posizionamento dev’essere riconosciuto dal t-RNA (attraverso codice/anticodice). Intervengono le transferasi, che portano al legame peptidico consumando GTP. Queste hanno Mg che guida la catalisi, avendo 2 interazioni verso l’enzima, e 2 verso i substrati, promuovendone la destabilizzazione sia dell’anidride (quello di GTP → GDP + Pi), sia dell’estere carbossilico (quello di t-RNA e del 1° amminoacido).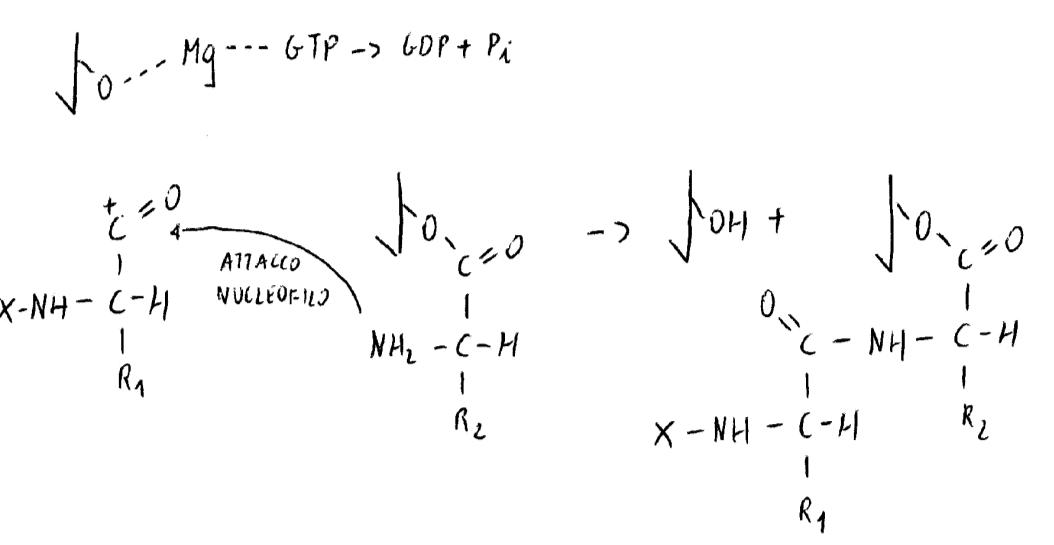
RNA della subunità maggiore ha 2 interazioni. Sul sito A c’è t-RNA che ha perso il 1° amminoacido trasportato. Sul secondo sito c’è una prima struttura peptidica. Il t-RNA del 2° amminoacido sta portando non un amminoacido, ma un dipeptide. L’energia conformazionale che si è resa disponibile con l’idrolisi di GTP, realizza cambiamenti nell’assetto conformazionale, trasformando A in U, e t-RNA viene dissociato. A valle del sito A si realizza una situazione favorevole all’ingresso del nuovo t-RNA con l’amminoacido specifico. Intervengono le altre due transferasi utilizzando GTP, una ha un sito per il t-RNA specifico che promuove l’idrolisi di GTP, usando l’energia conformazionale per il reciproco scorrimento tra ribosoma e t- RNA (è come se saltasse una tripletta). I fattori di allungamento agiscono sul nuovo sito A formato (interagisce solo quello che porta l’amminoacido riconosciuto attraverso codice/anticodice). C’è il sito P con un dipeptide e il nuovo sito A con il 3° amminoacido trasportato. La prima transferasi con Mg e GTP inizia una nuova destabilizzazione. Viene formato un peptide; quindi t-RNA è scarico (sito U) e si ha nel sito A una struttura peptidica. GTP viene idrolizzato e si ha un nuovo scorrimento della posizione del codice (tripletta). I siti A, P e U sono ora favorevoli per l’ingresso di un quarto t-RNA. Tutto si ripete finché non arriva il segnale d’arresto, man mano che il ribosoma scorre sul t-RNA.
La sintesi proteica avviene in forma multipla attraverso poliribosomi, dato che i peptidi possono essere molto lunghi. Ci sono segnali per il ribosoma che indicano fine lettura (sequenza di poliadenosina), e il ribosoma abbandona il t-RNA. Avviene avvolgimento e stabilizzazione (struttura secondaria, supersecondaria e terziaria), quindi trasferimento in compartimenti precisi. La metionina, marker di trasferimento, è il frammento d testa (un codice che indica la direzione verso il comparto cellulare). La metionina viene tagliata prima del completamento. La proteine chaperonine assestano la struttura al di sopra della primaria, la accettano e fanno gli avvolgimenti corretti.
Continua a leggere:
- Successivo: Il ciclo metabolico di Calvin
- Precedente: Ligasi Mn-dipendenti
Dettagli appunto:
-
Autore:
Marco Lazzara
[Visita la sua tesi: "Determinazione di arsenico mediante voltammetria di stripping anodico con elettrodo in oro"]
[Visita la sua tesi: "I laser a coloranti: fondamenti e stato dell'arte"]
- Università: Università degli Studi di Torino
- Facoltà: Scienze Matematiche, Fisiche e Naturali
- Corso: Chimica
- Esame: Biochimica
- Docente: Carlo Giunta
Altri appunti correlati:
- Biochimica
- Fondamenti di chimica
- Appunti di Biochimica
- Catecolamine e glucagone
- Chimica Analitica Strumentale (Spettroscopia)
Per approfondire questo argomento, consulta le Tesi:
- Analisi dei nitriti attraverso il loro impiego nell’industria alimentare e applicazione di un’indagine percettiva sui consumatori
- Meccanismi biochimici ed effetti sulla salute umana del licopene
- Studio dei processi di degrado su manufatti archeologici in ambiente marino sommerso e sperimentazione di procedure innovative per la conservazione in situ
- Bio-lubricant from the olive mill waste through a new biocatalyst immobilized on ZnFe2O4
- Rational Design and Synthesis of Novel Acadesine-Like Modulators of AMPK
Puoi scaricare gratuitamente questo appunto in versione integrale.